[本站讯]近日,控制科学与工程学院刘治平教授团队在国际学术期刊Genome Research在线发表了题为“Diffusion-based generation of gene regulatory network from scRNA-seq data with DigNet”的研究论文。本研究开发了一种名为DigNet的新型预训练计算框架,通过结合离散扩散生成模型和图嵌入方法,实现了从单细胞RNA测序数据到基因调控网络的端到端直接生成的新模式,显著提高了基因调控网络推断任务的精度与效率,为解析复杂生物分子网络、发现信号通路和寻找疾病生物标志物提供了有力的新工具。博士研究生王传远为第一作者,刘治平教授为通讯作者,山东大学为第一作者单位和通讯作者单位。
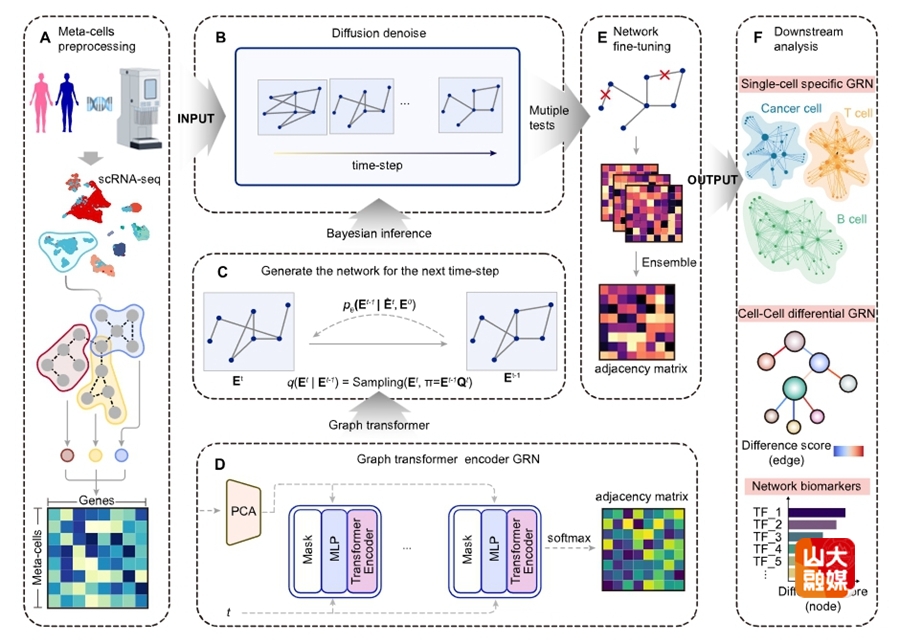
DigNet框架流程图
基因调控网络精确地编码了细胞内基因角色与功能之间的相互作用,从而决定了细胞的特异性。尽管历经了数十年的研究,从基因表达数据中逆向构建基因调控网络仍然面临巨大挑战,尤其是在构建能够精确匹配细胞和遗传背景特异性的基因调控网络时。为此,DigNet将网络推断任务分解为一系列具有马尔可夫特性的多步扩散过程。每一步都运用特定模型重建部分基因调控架构,以确保网络结构与基因表达谱之间的高度契合。这种生成模式不仅考虑了多个基因间的复杂调控关系,还着重关注了调控网络中的全局结构信息。此外,通过结合元细胞(Meta-cell)集成技术与非欧几里德离散空间建模,DigNet能够有效应对数据中的噪声问题并克服网络稀疏性的挑战。
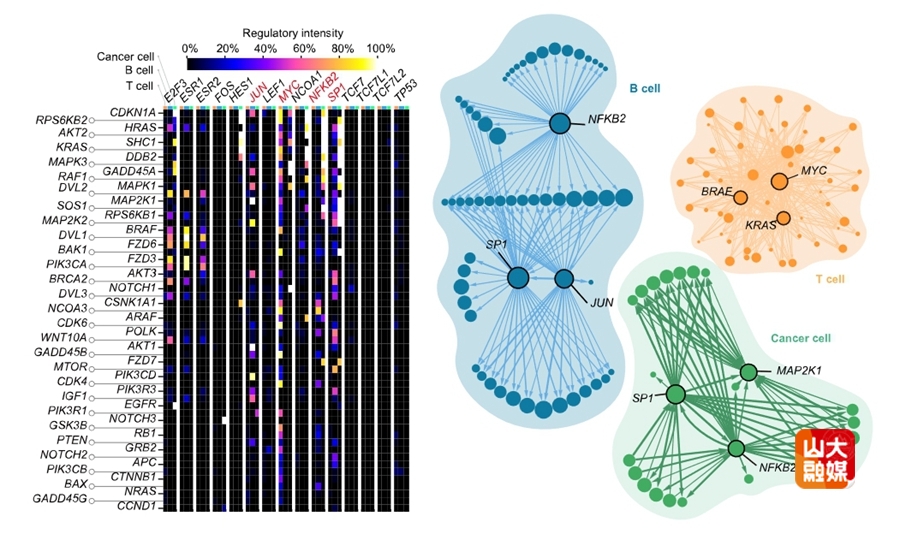
DigNet构建的人类乳腺癌免疫细胞特异性基因调控网络图
本研究在多个基准数据集上验证了DigNet方法的有效性。实验结果表明,DigNet在基因调控网络推断精度方面超越了现有方法,达到了当前最高水平(state-of-the-art, SOTA)。同时,DigNet具有捕捉全局网络结构的能力,能够解析多个基因间的复杂调控关系,并直接生成调控网络结构。作为案例研究,该方法揭示了人类乳腺癌免疫细胞中的复杂调控机制,成功构建了免疫细胞特异性基因调控网络。通过重新确认已知的关键调控关系并筛选出之前未知的候选调控作用,DigNet揭示了细胞功能特异性的调控关系,展示了其在挖掘基于网络的生物标志物方面的实用价值,例如发现了SP1、SOS1、NFKB2和AKT2等基因的重要作用。这些新发现的差异调控相互作用为深入理解乳腺癌免疫细胞反应机制、探索新的治疗靶点提供了创新的方法与视角。
上述研究工作得到了国家重点研发计划、国家自然科学基金重大研究计划和山东大学集成攻关培育项目的大力支持。