
[本站讯]5月23日,mBio期刊在线发表了山东大学微生物改造技术全国重点实验室王海龙教授课题组的研究论文“Programming virulent bacteriophages by developing a multiplex genome engineering method”。王海龙教授为论文通讯作者,博士研究生张海林为论文第一作者。山东大学微生物改造技术全国重点实验室为第一完成单位和通讯作者单位。
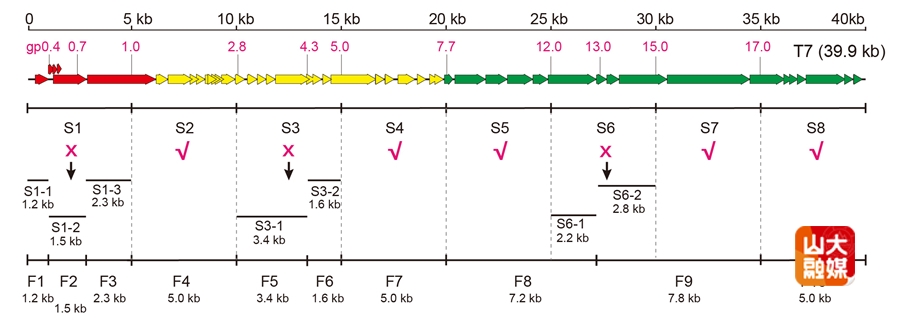
图1 SMART技术的噬菌体基因组拆分策略
噬菌体作为一种专门侵染并杀死细菌的病毒,在减少或替代抗生素治疗细菌感染方面发挥重要作用。天然噬菌体存在杀菌效率低、杀菌谱窄等缺陷,这促使人们使用基因编辑技术改造天然噬菌体创建功能优化的工程噬菌体。然而,烈性噬菌体在数分钟内迅速裂解宿主,仅留下极其狭窄的基因编辑窗口期。因此,在细菌宿主中高效地完成噬菌体基因组编辑具有挑战性。该论文开发了一种可对烈性噬菌体进行多重基因组编辑的SMART技术(Splitting,Modifying,Assembling, andRebooTing),以有效规避基因编辑窗口期短的限制。SMART技术首先将噬菌体基因组拆分成多个片段(图1),并克隆至单拷贝细菌人工染色体载体中(Splitting),避免噬菌体基因对克隆宿主毒性。然后,通过课题组前期开发的Red-ccdB无痕定点突变技术(Nucleic Acids Research,2014, 42, e37),对所有目的片段进行改造(Modifying)。最后,利用课题组前期开发的ExoCET多片段组装技术(Nucleic Acids Research,2018, 46, e28),在细菌宿主细胞内完成改造后噬菌体基因组的组装与激活(Assembling and Rebooting),最终获得工程噬菌体。
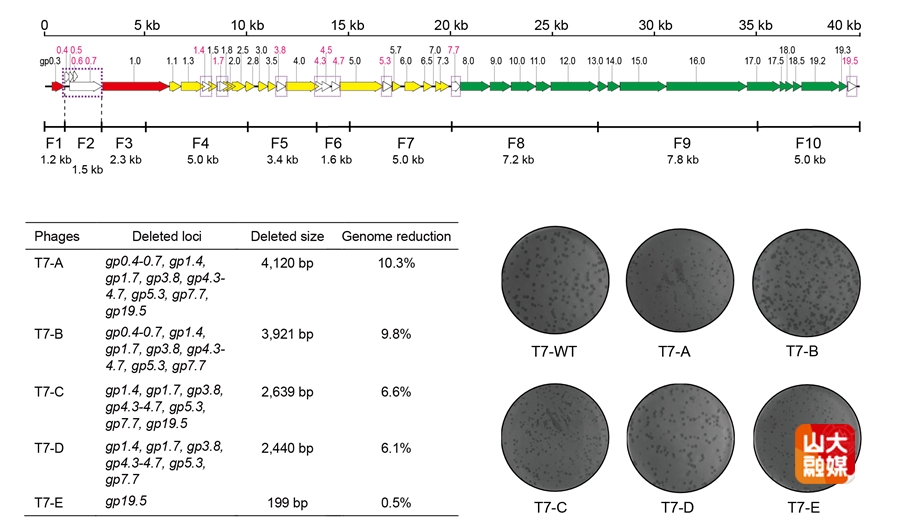
图2 利用SMART技术对噬菌体基因组进行多位点编辑和构建底盘噬菌体
该论文利用SMART技术,将39.9 kb的T7大肠杆菌噬菌体基因组拆分为10个可克隆的片段,删除基因组中8个非必需基因区域,评估非必需基因敲除对噬菌体活性的影响,最终获得基因组减少10%并保持高裂解活性的底盘噬菌体(图2)。随后,进一步测试底盘噬菌体装载外源基因的容量以及外源基因在基因组不同位点的表达水平,鉴定出多个外源基因表达水平较高的位点。
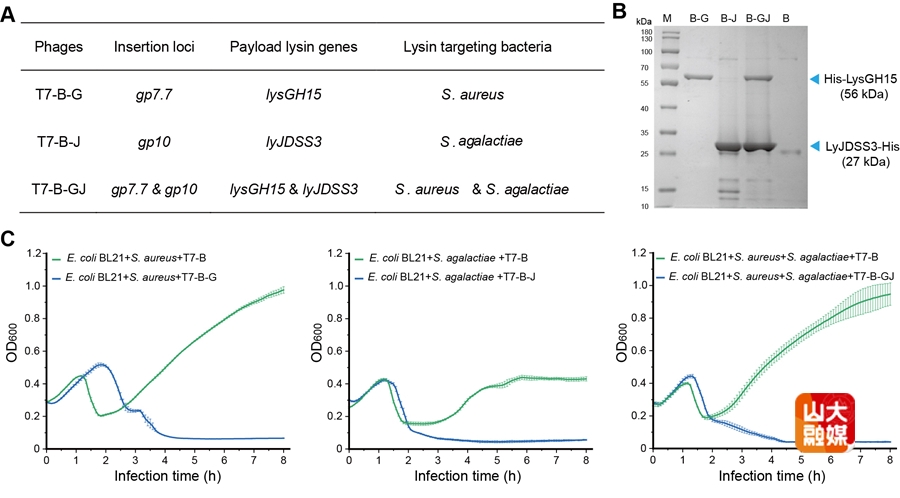
图3 SMART技术构建的工程噬菌体裂解大肠杆菌、金黄色葡萄球菌和无乳链球菌
为了扩展T7大肠杆菌噬菌体的裂解谱,将来自金黄色葡萄球菌和链球菌噬菌体的裂解酶基因装载至T7底盘噬菌体基因组中,构建工程噬菌体。体外共培养实验表明,工程噬菌体在裂解大肠杆菌宿主过程中可原位释放两种异源裂解酶,并能有效地裂解金黄色葡萄球菌和无乳链球菌(图3)。该研究构建的工程噬菌体可跨越种属的特异性限制,拓宽其杀菌谱,为治疗多种病原菌引起的复合感染提供一种思路。
基于质粒在细菌复制的高保真性,SMART噬菌体基因组编辑技术构建的工程噬菌体,只需对改造后的目的基因区域进行测序验证,无需对噬菌体全基因组测序,即可进一步分析基因型-表型关系。而且,SMART技术可以对多个基因位点同时实施编辑,后期重组噬菌体筛选过程简单易行,无野生型噬菌体干扰。SMART技术丰富了噬菌体基因组编辑工具箱,有利于工程噬菌体的高效创建。
本研究得到了山东大学教授张友明、上海交通大学教授严亚贤的支持,以及国家重点研发计划、山东省自然科学基金、微生物改造技术全国重点实验室揭榜挂帅项目、微生物改造技术全国重点实验室生命科学领域校内联合项目等项目资助。
