[本站讯]近日,山东大学软件学院吴昊副教授团队在细胞生命周期预测方面取得新进展,相关成果以“scHiCyclePred: a deep learning framework for predicting cell cycle phases from single-cell Hi-C data using multi-scale interaction information”为题在线发表在Nature旗下的生物信息学国际权威学术期刊Communications Biology。吴昊副教授和加拿大麦吉尔大学医学院研究员丁俊为论文通讯作者,山东大学软件学院为第一作者和通讯作者单位。

单细胞高通量染色质构象捕获(scHi-C)技术的出现为研究细胞周期阶段与染色质三维(3D)结构之间的复杂关系提供了前所未有的机会。然而,基于scHi-C数据准确预测细胞周期阶段仍然是一个艰巨的挑战。因此,一些研究的重点是构建细胞周期的伪轨迹序列。例如,Nagano等从小鼠胚胎干细胞中获得了不同细胞周期的scHi-C数据,并利用机器学习算法计算细胞的相关指标,进而推断出细胞的伪轨迹序列;Ye等人提出了CIRCLET方法,该方法基于多个特征集的组合,通过计算K-最近邻图来重建细胞周期伪轨迹序列。但是,尽管从scHi-C数据构建细胞周期的伪轨迹序列具有一定的应用潜力,但由于缺乏包含每个细胞周期阶段细胞数量信息的通用数据集,因此无法直接预测出单个细胞的细胞周期阶段。此外,CIRCLET只能重建出细胞周期的伪轨迹序列,不能直接预测单个细胞的细胞周期,尤其是处于周期边界的细胞。同时,CIRCLET的实现并不是完全自动化的,需要设置细胞伪轨迹序列的起点,这通常需要一定程度的经验和试错调整。
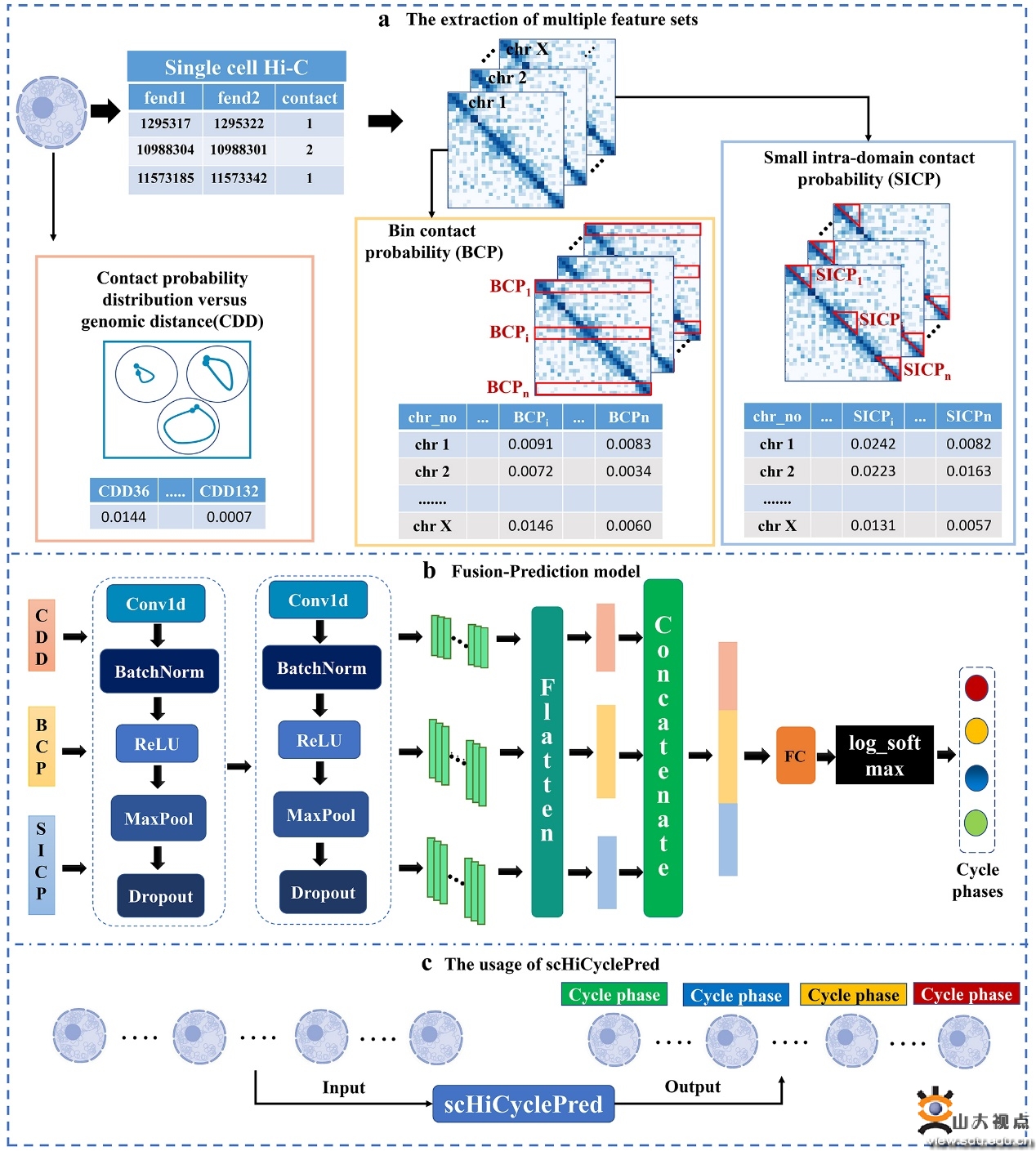
为了解决从scHi-C数据预测细胞具体周期阶段面临的挑战,研究针对单细胞Hi-C数据进行深度分析,构建了三种描述细胞特征的方法CDD、BCP和SICP,从不同角度全面地获取细胞的特征刻画,然后提出了一个基于深度学习计算模型scHiCyclePred。该模型不仅结合了多尺度交互信息提取三维基因组染色质结构特征,而且对细胞周期中的三维染色质动力学和基因特征进行了深度分析和刻画,为细胞周期动力学特征提供了更全面的理解。scHiCyclePred不仅可以精确地预测细胞周期阶段,而且提供了对细胞生物学结构的深入剖析,该研究有望在复杂疾病研究中发挥重要作用,促进复杂疾病研究进展。
